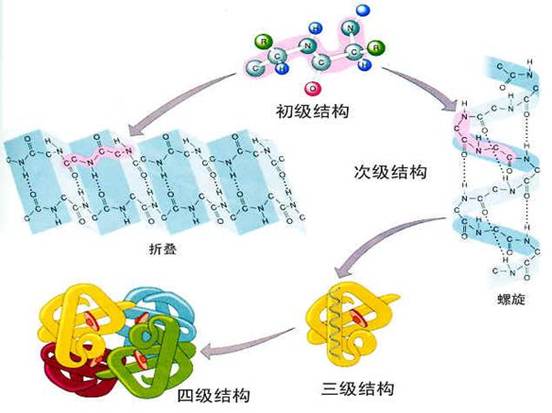
蛋白質結構是指蛋白質分子的空間結構。蛋白質主要由碳、氫、氧、氮等化學元素組成,是一類重要的生物大分子,所有蛋白質都是由20種不同胺基酸連線形成的多聚體,在形成蛋白質後,這些胺基酸又被稱為殘基。
蛋白質和多肽之間的界限並不是很清晰,有人基於發揮功能性作用的結構域所需的殘基數認為,若殘基數少於40,就稱之為多肽或肽。要發揮生物學功能,蛋白質需要正確摺疊為一個特定構型,主要是通過大量的非共價相互作用(如氫鍵,離子鍵,范德華力和疏水作用)來實現;此外,在一些蛋白質(特別是分泌性蛋白質)摺疊中,二硫鍵也起到關鍵作用。為了從分子水平上了解蛋白質的作用機制,常常需要測定蛋白質的三維結構。由研究蛋白質結構而發展起來了結構生物學,採用了包括X射線晶體學、核磁共振等技術來解析蛋白質結構。
一定數量的殘基對於發揮某一生物化學功能是必要的;40-50個殘基通常是一個功能性結構域大小的下限。蛋白質大小的範圍可以從這樣一個下限一直到數千個殘基。目前估計的蛋白質的平均長度在不同的物種中有所區別,一般約為200-380個殘基,而真核生物的蛋白質平均長度比原核生物長約55%。更大的蛋白質聚合體可以通過許多蛋白質亞基形成;如由數千個肌動蛋白分子聚合形成蛋白纖維。
基本介紹
發現歷史
結構種類
一級結構
蛋白質的一級結構決定了蛋白質的二級、三級等高級結構,成百億的天然蛋白質各有其特殊的生物學活性,決定每一種蛋白質的生物學活性的結構特點,首先在於其肽鏈的胺基酸序列,由於組成蛋白質的20種胺基酸各具特殊的側鏈,側鏈基團的理化性質和空間排布各不相同,當它們按照不同的序列關係組合時,就可形成多種多樣的空間結構和不同生物學活性的蛋白質分子。
蛋白質分子的多肽鏈並非呈線形伸展,而是摺疊和盤曲構成特有的比較穩定的空間結構。蛋白質的生物學活性和理化性質主要決定於空間結構的完整,因此僅僅測定蛋白質分子的胺基酸組成和它們的排列順序並不能完全了解蛋白質分子的生物學活性和理化性質。例如球狀蛋白質(多見於血漿中的白蛋白、球蛋白、血紅蛋白和酶等)和纖維狀蛋白質(角蛋白、膠原蛋白、肌凝蛋白、纖維蛋白等),前者溶於水,後者不溶於水,顯而易見,此種性質不能僅用蛋白質的一級結構的胺基酸排列順序來解釋。
蛋白質的空間結構就是指蛋白質的二級、三級和四級結構。
二級結構
1.肽鍵平面(或稱醯胺平面,amide plane)。
Pauling等人對一些簡單的肽及胺基酸的醯胺等進行了X線衍射分析,從一個肽鍵的周圍來看,得知:
(1)肽鍵中的C-N鍵長0.132nm,比相鄰的N-C單鍵(0.147nm)短,而較一般C=N雙鍵(0.128nm)長,可見,肽鍵中-C-N-鍵的性質介於單、雙鍵之間,具有部分雙鍵的性質,因而不能旋轉,這就將固定在一個平面之內。
(2) 肽鍵的C及N周圍三個鍵角之和均為360°,說明都處於一個平面上,也就是說六個原子基本上同處於一個平面,這就是肽鍵平面。肽鏈中能夠旋轉的只有α碳原子所形成的單鍵,此單鍵的旋轉決定兩個肽鍵平面的位置關係,於是肽鍵平面成為肽鏈盤曲摺疊的基本單位。
(3) 肽鍵中的C-N既具有雙鍵性質,就會有順反不同的立體異構,已證實處於反位。
2.蛋白質主鏈構象的結構單元
1)α-螺旋Pauling等人對α-角蛋白(α-keratin)進行了X線衍射分析,從衍射圖中看到有0.5~0.55nm的重複單位,故推測蛋白質分子中有重複性結構,並認為這種重複性結構為α-螺旋(α-helix).
α-螺旋的結構特點如下:
①多個肽鍵平面通過α-碳原子旋轉,相互之間緊密盤曲成穩固的右手螺旋。
②主鏈呈螺旋上升,每3.6個胺基酸殘基上升一圈,相當於0.54nm,這與X線衍射圖符合。
③相鄰兩圈螺旋之間借肽鍵中C=O和H桸形成許多鏈內氫健,即每一個胺基酸殘基中的NH和前面相隔三個殘基的C=O之間形成氫鍵,這是穩定α-螺旋的主要鍵。
④肽鏈中胺基酸側鏈R,分布在螺旋外側,其形狀、大小及電荷影響α-螺旋的形成。酸性或鹼性胺基酸集中的區域,由於同電荷相斥,不利於α-螺旋形成;較大的R(如苯丙氨酸、色氨酸、異亮氨酸)集中的區域,也妨礙α-螺旋形成;脯氨酸因其α-碳原子位於五元環上,不易扭轉,加之它是亞胺基酸,不易形成氫鍵,故不易形成上述α-螺旋;甘氨酸的R基為H,空間占位很小,也會影響該處螺旋的穩定。
2)β-片層結構Astbury等人曾對β-角蛋白進行X線衍射分析,發現具有0.7nm的重複單位。如將毛髮α-角蛋白在濕熱條件下拉伸,可拉長到原長二倍,這種α-螺旋的X線衍射圖可改變為與β-角蛋白類似的衍射圖。說明β-角蛋白中的結構和α-螺旋拉長伸展後結構相同。兩段以上的這種摺疊成鋸齒狀的肽鏈,通過氫鍵相連而平行成片層狀的結構稱為β-片層(β-pleated sheet)結構或稱β-折迭。
β-片層結構特點是:
①是肽鏈相當伸展的結構,肽鏈平面之間摺疊成鋸齒狀,相鄰肽鍵平面間呈110°角。胺基酸殘基的R側鏈伸出在鋸齒的上方或下方。
②依靠兩條肽鏈或一條肽鏈內的兩段肽鏈間的C=O與N-H形成氫鍵,使構象穩定。
③兩段肽鏈可以是平行的,也可以是反平行的。即前者兩條鏈從“N端”到“C端”是同方向的,後者是反方向的。β-片層結構的形式十分多樣,正、反平行能相互交替。
④平行的β-片層結構中,兩個殘基的間距為0.65nm;反平行的β-片層結構,則間距為0.7nm.
3)β-轉角
蛋白質分子中,肽鏈經常會出現180°的回折,在這種回折角處的構象就是β-轉角(β-turn或β-bend)。β-轉角中,第一個胺基酸殘基的C=O與第四個殘基的N-H之間形成氫鍵,從而使結構穩定。
4)無規捲曲
沒有確定規律性的部分肽鏈構象,肽鏈中肽鍵平面不規則排列,屬於鬆散的無規捲曲(random coil)。
超二級結構
結構域(domain)也是蛋白質構象中二級結構與三級結構之間的一個層次。在較大的蛋白質分子中,由於多肽鏈上相鄰的超二級結構緊密聯繫,形成二個或多個在空間上可以明顯區別它與蛋白質亞基結構的區別。一般每個結構域約由100-200個胺基酸殘基組成,各有獨特的空間構象,並承擔不同的生物學功能。如免疫球蛋白(IgG)由12個結構域組成,其中兩個輕鏈上各有2個,兩個重鏈上各有4個;補體結合部位與抗原結合部位處於不同的結構域。一個蛋白質分子中的幾個結構域有的相同,有的不同;而不同蛋白質分子之間肽鏈中的各結構域也可以相同。如乳酸脫氫酶、3-磷酸甘油醛脫氫酶、蘋果酸脫氫酶等均屬以NAD+為輔酶的脫氫酶類,它們各自由2個不同的結構域組成,但它們與NAD+結合的結構域構象則基本相同。
三級結構
現也有認為蛋白質的三級結構是指蛋白質分子主鏈摺疊盤曲形成構象的基礎上,分子中的各個側鏈所形成一定的構象。側鏈構象主要是形成微區(或稱結構域domain)。對球狀蛋白質來說,形成疏水區和親水區。親水區多在蛋白質分子表面,由很多親水側鏈組成。疏水區多在分子內部,由疏水側鏈集中構成,疏水區常形成一些“洞穴”或“口袋”,某些輔基就鑲嵌其中,成為活性部位。
具備三級結構的蛋白質從其外形上看,有的細長(長軸比短軸大10倍以上),屬於纖維狀蛋白質(fibrous protein),如絲心蛋白;有的長短軸相差不多基本上呈球形,屬於球狀蛋白質(globular protein),如血漿清蛋白、球蛋白、肌紅蛋白,球狀蛋白的疏水基多聚集在分子的內部,而親水基則多分布在分子表面,因而球狀蛋白質是親水的,更重要的是,多肽鏈經過如此盤曲後,可形成某些發揮生物學功能的特定區域,例如酶的活性中心等。
四級結構
一種蛋白質中,亞基結構可以相同,也可不同。如菸草斑紋病毒的外殼蛋白是由2200個相同的亞基形成的多聚體;正常人血紅蛋白A是兩個α亞基與兩個β亞基形成的四聚體;天冬氨酸氨甲醯基轉移酶由六個調節亞基與六個催化亞基組成。有人將具有全套不同亞基的最小單位稱為原聚體(protomer),如一個催化亞基與一個調節亞基結合成天冬氨酸氨甲醯基轉移酶的原聚體。
某些蛋白質分子可進一步聚合成聚合體(polymer)。聚合體中的重複單位稱為單體(monomer),聚合體可按其中所含單體的數量不同而分為二聚體、三聚體……寡聚體(oligomer)和多聚體(polymer)而存在,如胰島素(insulin)在體內可形成二聚體及六聚體。
作用
套用
在結構基因組學中的套用
在藥物設計中的套用
在蛋白質設計中的套用
組成
化學組成
分子組成
元素組成
功能分類
肽鍵
肽鍵 | 平均長度 | 單鍵 | 平均長度 | 氫鍵 | 平均長度(±30) |
Cα - C | 153 pm | C - C | 154 pm | O-H --- O-H | 280 pm |
C - N | 133 pm | C - N | 148 pm | N-H --- O=C | 290 pm |
N - Ca | 146 pm | C - O | 143 pm | O-H --- O=C | 280 pm |
側鏈構象
類型
蛋白質摺疊
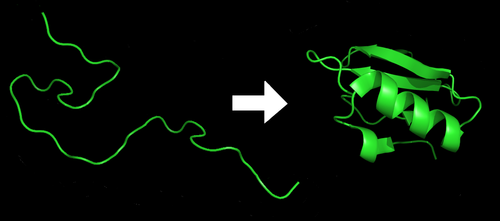 蛋白質摺疊前後
蛋白質摺疊前後結構分類
結構測定
解析度(埃) | 結構中可能出現的問題 |
>4.0 | 單個原子坐標無意義 |
3.0 - 4.0 | 整體摺疊可能是正確的,但很可能有錯誤存在。很多側鏈擺放位置不正確。 |
2.5 - 3.0 | 整體摺疊基本是正確的,除了位於結構表面的一些環狀結構可能沒有正確建模。長側鏈的極性殘基(Lys、Glu、Gln等)和小側鏈殘基(Ser、Val、Thr等)的側鏈擺放位置有可能不正確。 |
2.0 - 2.5 | 與2.5 - 3.0類似,只是出現錯誤的情況更少。可以明顯觀察到水分子和小配基。 |
1.5 - 2.0 | 側鏈擺放位置基本無誤,甚至一些小的錯誤也可以被檢測到。整體摺疊,包括位於結構表面的環狀結構,基本不可能出現錯誤。 |
0.5 - 1.5 | 在這一解析度下,一般不會有結構錯誤。側鏈異構體庫和立體幾何研究都是利用這一解析度範圍內的結構來進行的。 |
結構預測
- 二級結構預測
- 三級結構預測
- 同源建模:需要有同源的蛋白三級結構為基礎進行預測。
- “從頭開始”(Ab initio):只需要蛋白質序列即可進行結構預測。由於運算量大,需要有超級計算機來進行,或採用分散式計算,如Rosetta@home等。