Sun L, Zeng X, Yan C, Sun X, Gong X, Rao Y, Yan N. Crystal structure of a bacterial homologue of glucose transporters GLUT1–4. Nature, 2012; 490:361–366.
Yin P, Deng D, Yan C, Pan X, Xi JJ, Yan N*, Shi Y*.Specific DNA-RNA Hybrid Recognition by TAL Effectors. Cell Rep. 2012; Sep 26. Epub ahead of print (co-corresponding authors)
Deng D, Yin P, Yan C, Pan X, Gong X, Qi S, Xie T, Mahfouz M, Zhu JK, Yan N*, Shi Y*. Recognition of methylated DNA by TAL effectors. Cell Res. 2012;22(10):1502-4. (co-corresponding authors)
Zhang X, Ren W, DeCaen P, Yan C, Tao X, Tang L, Wang J, Hasegawa K, Kumasaka T, He J, Wang J, Clapham DE, Yan N. Crystal structure of an orthologue of the NaChBac voltage-gated sodium channel. Nature. 2012;486(7401):130-4.
Tian Xie, Ruobing Ren, Yuan-yuan Zhang, Yuxuan Pang, Chuangye Yan, Xinqi Gong, Yuan He, Wenqi Li, Di Miao, Qi Hao, Haiteng Deng, Zhixin Wang, Jia-Wei Wu and Nieng Yan. Molecular Mechanism for Inhibition of a Critical Component in the Arabidopsis thaliana Abscisic Acid Signal Transduction Pathways, SnRK2.6, by Protein Phosphatase ABI1. J Biol Chem. 2012; 287:794-802.
Dong Deng, Chuangye Yan, Xiaojing Pan, Magdy Mahfouz, Jiawei Wang, Jian-Kang Zhu, Yigong Shi*, and Nieng Yan* Structural basis for the specific recognition of DNA by TAL effectors. Science. 2012; 335(6069):720-3, Epub: 2012 Jan 5. (* indicates corresponding authors).
Hao Q, Yin P, Li W, Wang L, Yan C, Lin Z, Wu JZ, Wang J, Yan SF, Yan N. The molecular basis of ABA-independent inhibition of PP2Cs by a subclass of PYL proteins.Mol Cell. 2011;42(5):662-72.
Lu F, Li S, Jiang Y, Jiang J, Fan H, Lu G, Deng D, Dang S, Zhang X, Wang J, Yan N. Structure and mechanism of the uracil transporter UraA. Nature. 2011;472(7342):243-6.
Dang S, Sun L, Huang Y, Lu F, Liu Y, Gong H, Wang J, Yan N. Structure of a fucose transporter in an outward-open conformation. Nature. 2010;467(7316):734-8.
Yuan X, Yin P, Hao Q, Yan C, Wang J, Yan N. Single amino acid alteration between valine and isoleucine determines the distinct pyrabactin selectivity by PYL1 and PYL2. J Biol Chem. 2010; 285(37):28953-8.
Hao Q, Yin P, Yan C, Yuan X, Li W, Zhang Z, Liu L, Wang J, Yan N. Functional mechanism of the abscisic acid agonist pyrabactin. J Biol Chem. 2010;285(37):28946-52.
Qi S, Pang Y, Hu Q, Liu Q, Li H, Zhou Y, He T, Liang Q, Liu Y, Yuan X, Luo G, Li H, Wang J,Yan N*,Shi Y*. Crystal structure of the Caenorhabditis elegans apoptosome reveals an octameric assembly of CED-4. Cell. 2010;141(3):446-57. (co-corresponding authors)
Wang Y, Huang Y, Wang J, Cheng C, Huang W, Lu P, Xu YN, Wang P, Yan N*, Shi Y*. Structure of the formate transporter FocA reveals a pentameric aquaporin-like channel. Nature. 2009;462(7272):467-72. (co-corresponding authors)
Yin P, Fan H, Hao Q, Yuan X, Wu D, Pang Y, Yan C, Li W, Wang J, Yan N. Structural insights into the mechanism of abscisic acid signaling by PYL proteins. Nat Struct Mol Biol. 2009;16(12):1230-6.
Prior to 2007
Yan N, Shi Y. Allosteric activation of a bacterial stress sensor. Cell. 2007;131(3):441-3.
Wu Z*, Yan N*, Feng L*, Oberstein A, Yan H, Baker RP, Gu L, Jeffrey PD, Urban S, Shi Y. Structural analysis of a rhomboid family intramembrane protease reveals a gating mechanism for substrate entry.Nat Struct Mol Biol. 2006;13(12):1084-91.(co-first authors)
Yan N, Huh JR, Schirf V, Demeler B, Hay BA, Shi Y. Structure and activation mechanism of the Drosophila initiator caspase Dronc. J Biol Chem. 2006;281(13):8667-74.
Yan N, Xu Y, Shi Y. 2:1 Stoichiometry of the CED-4-CED-9 complex and the tetrameric CED-4: insights into the regulation of CED-3 activation. Cell Cycle. 2006;5(1):31-4.
Yan N, Shi Y. Mechanisms of apoptosis through structural biology. Annu Rev Cell Dev Biol. 2005; 21:35-56.
Yan N, Chai J, Lee ES, Gu L, Liu Q, He J, Wu JW, Kokel D, Li H, Hao Q, Xue D, Shi Y. Structure of the CED-4-CED-9 complex provides insights into programmed cell death in Caenorhabditis elegans. Nature. 2005; 437(7060):831-7.
Yan N, Gu L, Kokel D, Chai J, Li W, Han A, Chen L, Xue D, Shi Y. Structural, biochemical, and functional analyses of CED-9 recognition by the proapoptotic proteins EGL-1 and CED-4. Mol Cell. 2004; 15(6):999-1006.
Yan N, Wu JW, Chai J, Li W, Shi Y. Molecular mechanisms of DrICE inhibition by DIAP1 and removal of inhibition by Reaper, Hid and Grim. Nat Struct Mol Biol. 2004;11(5):420-8.
Yan N, Shi Y. Histone H1.2 as a trigger for apoptosis. Nat Struct Biol. 2003;10(12):983-5.
Chai J*, Yan N*, Huh JR, Wu JW, Li W, Hay BA, Shi Y. Molecular mechanism of Reaper-Grim-Hid-mediated suppression of DIAP1-dependent Dronc ubiquitination. Nat Struct Biol. 2003;10(11):892-8. (co-first authors)
 生活中的顏寧教授
生活中的顏寧教授 顏寧
顏寧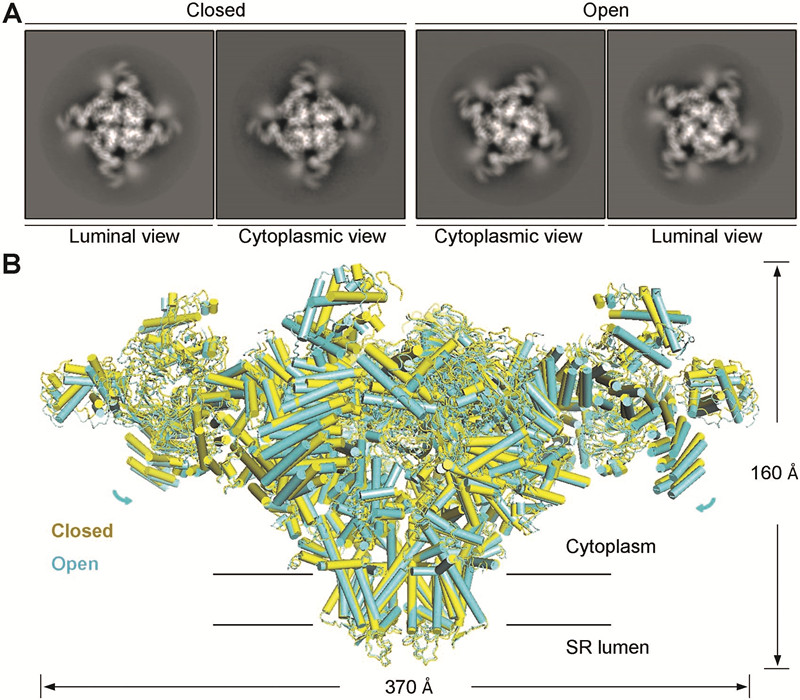 2016年9月-Science-關閉及開放構象的RyR2
2016年9月-Science-關閉及開放構象的RyR2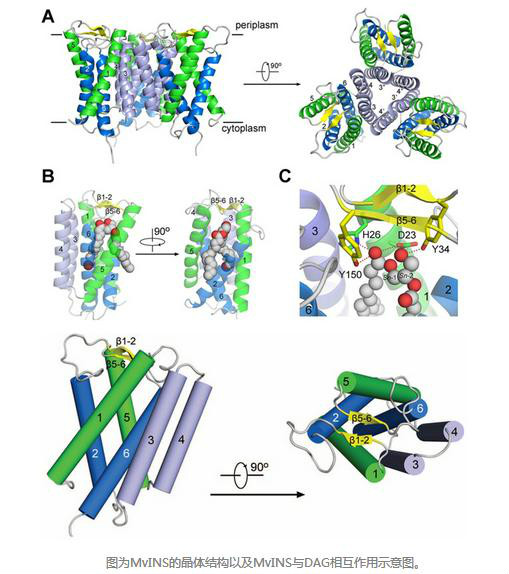 解析·蛋白結構與功能
解析·蛋白結構與功能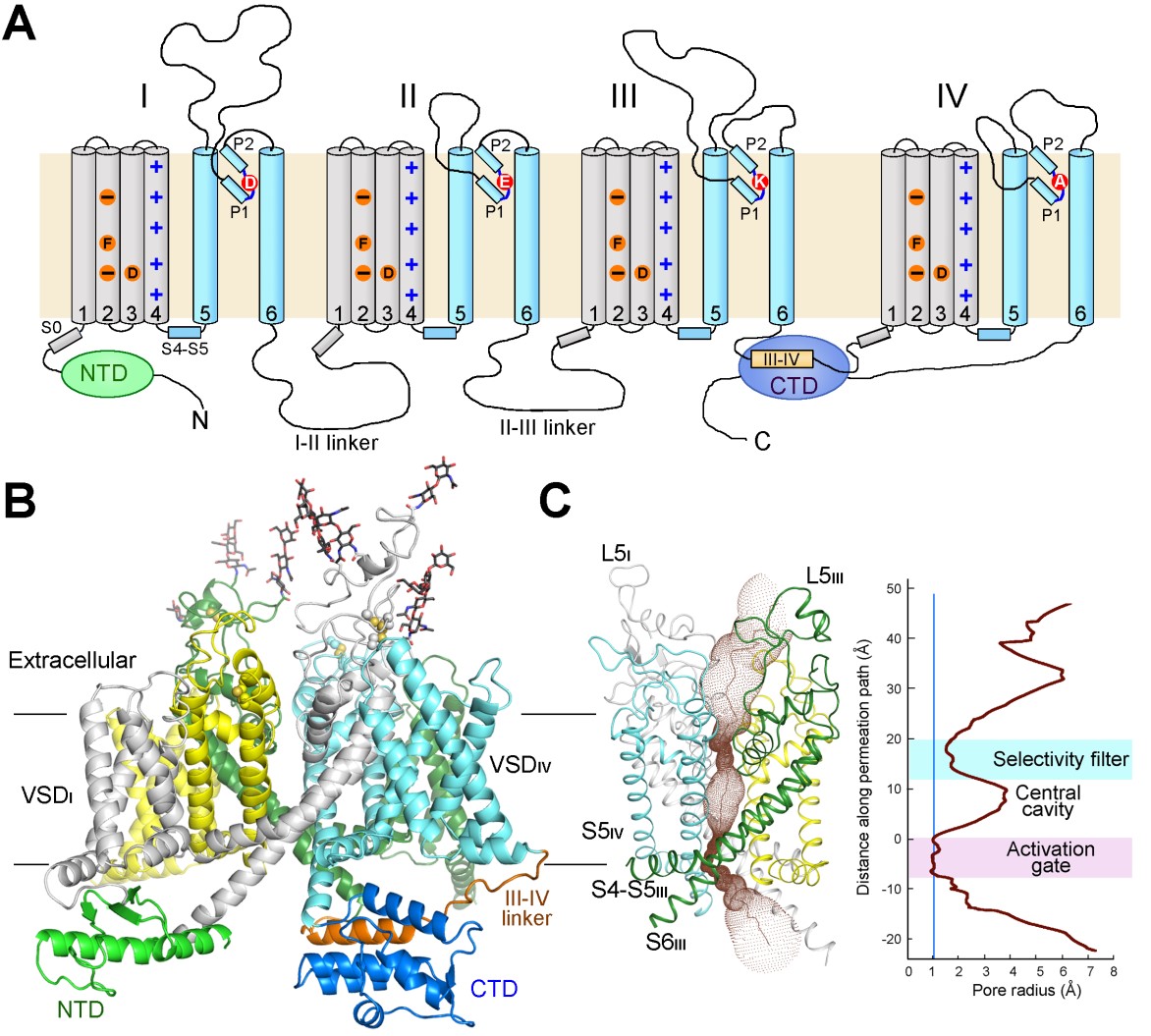 2017年2月,真核生物電壓門控鈉離子通道的拓撲圖和三維電鏡結構
2017年2月,真核生物電壓門控鈉離子通道的拓撲圖和三維電鏡結構
 顏寧
顏寧