由來 方法 三點測交
摩爾根的學生
斯特蒂文特 (Sturtevant)想出了一個的辦法進行
遺傳作圖 ,那就是
三點測交 (three-point test cross)。從幾何知識上知道,要證明a、b、c三點共線,可以通過三點之間的距離加以證明,當ab+bc=ac時三點在一條直線上。斯特蒂文特借鑑了這一思路,選擇了六個
性連鎖基因 進行果蠅的雜交實驗,我們以紅眼
V (Vermilion eyes)、翅無脈
Cv (crossveinless)和截翅
Ct (cut,or snipped wing edges)三個基因為例,雜交及結果如下。
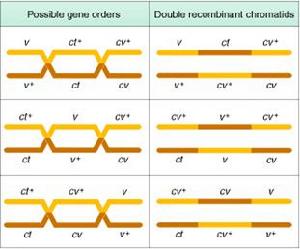
遺傳作圖 根據計算的結果,可以繪出
V 、Cv 、Ct 的遺傳圖。但是我們會發現
V -Ct (13.2 m.u)和
Ct -Cv (6.5 m.u)之和為19.7 m.u,大於
V -Cv 的距離(18.5 m.u),因為18.5 m.u這個數據是
V -Cv 的
重組值 ,即從
V 和
Cv 這兩個標記的重組中直接得來的,這兩點中如果發生雙交換的話,因不會產生重組而無法計算,易被漏掉,但從Cv-
Ct 和
V -Cv 的重組中可以看到確實發生了兩次
雙交換 ,交換率為0.6 %×2=1.2%,如果把漏掉的雙交換值加進去則:
(V -Cv )=(Cv -Ct )+(V -Cv )
18.5 m.u+1.2 m.u=6.5 m.u+13.2 m.u=19.7 m.u
因此我們可用下面的公式來表示a-b之間的重組值和雙交換的關係:
ab =ac +cb -2 (ac )(cb )
ab 是a 和b 兩個基因之間的重組值。c 是a 與b 之間的一個基因,ac、cb 表
示a 和c 、c 和b 的重組值,( ac )(cb ) 表示雙交換值。
形態學標記 最初的
遺傳學圖 是在20世紀初對
果蠅 等生物構建的,使用基因作為際記:許多年之後人們才認識到基因是
DNA 分子的片段。而在當時,基因被認為是能將可
遺傳 的性狀從親子傳遞到後代的抽象實體一個遺傳性狀必須以兩種替換形式或表型(phenotype)存在才能用於遺傳學分析。如
孟德爾 首先研究的豌豆莖的高或矮。每種表型是由相應基因的不同
等位基因 (allele)所決定的。起切只有那些能通過視覺區分的基因表型能用於研究。比如,第一張果蠅遺傳圖顯示了負責身體顏色、眼睛顏色、翅膀形態等基因的位置,這些表型都可在低倍顯微鏡下或肉眼觀察果蠅而看到。
遺傳作圖 早期尚覺得這種方法很精細,但遺傳學家們很快就發現,只有有限的幾種可見表型的遺傳可用於研究,而在許多情況下,由於不止一個基因影響一個物理特徵,分析起來並不大容易。例如,到1922年,有超過50個基因被定位在4條果蠅染色體上、而其中9個基因負責眼睛的顏色,想在此領域有所貢獻的每一個初涉者必須首先學會辨別果蠅眼睛的顏色是紅、淡紅、朱紅、朽榴石色、
康乃馨 色、肉桂色、深褐色、猩紅或深紅色。因此,為了使基因圖更加全而,有必要找到一些比可見的
性狀 更多、更明確而且更簡單的性狀。
生化標記 以上問題的解決方案之一是套用
生物 化學方法來區分表型。這對於
微生物 與
人類 這兩種生物尤為重要,
細菌 與
酵母 等微生物只有為數很少的可見性狀,因此這類生物的基因作圖只能依賴於少數的生化表型。人類雖然有可見的性狀持征,但以
血液 分型為代表的生化表型研究從20世紀20午代就開始了。
遺傳作圖 血液分型研究不僅包括如ABO系統的標準
血型 ,還有
血清蛋白 以及人類白細胞抗原〔HLA系統)等免疫蛋白的
等位基因 可變體。這些標記相對於可見表型的一個巨人優點是其相關基因往往為
復等位基因 (multiple allele)。例如,HLA—DRBI基因至少有59個等位基因,而HLA- B至少有60個。這正是與人類作圖相關的。與在果蠅或小鼠等生物中建立的雜交實驗不同,人類基因遺傳的數據只能通過檢查—個家族中各成員的表型來獲得。如果對所研究的基因而言,所有的家族成員都為純合子,就得不到有用的信息。這對於只有兩個等位基因的基因較常見,因為婚配可偶然地發生於同一個等位基因純合子個體之間。而當所研究的基因有60個而不是2個等位基因時,這就很少見了。
簡單的說:形態標記和生化標記的局限性在於多態性太低,可標記的位點太少。這是他們共同的致命弱點。
DNA分子標記 DNA 分子標記大多是以DNA片段電泳譜帶形式表現的。依其遺傳特性可分為顯性和共顯性標記2種;依多態性檢測手段可分為以Southern雜交技術為核心的
分子標記 和以PCR技術為核心的分子標記;根據在基因組中出現的頻率,又可分為低拷貝序列和重複序列標記。
遺傳作圖 限制性片段多態性
RFILP是第—種用於研究的DNA標記。限制性
核酸 內切酶是一種在特定序列上切割DNA分子的
酶 ,用它處理一個DNA分子時,即產生限制片段。這種序列特異性意味著用一種限制酶處理一種DNA分子總會產生同樣的片段。但對於基因組DNA來說,並不總是這樣。這是因為有些限制位點具多態性,以兩種等位形式存在。一種等位形式有正確的限制位點序列,能被酶切開;另一等位形式的序列有改變,從而該限制位點不能被識別。後者的結果是在
核酸內切酶 處理後,兩個相鄰的限制片段仍然連線在一起,從而導致了長度多態性(如圖1)。這就是一個RFLP的例子。如同用基因作為標記一樣,RFLP在基因組圖譜上的位置可以通過追蹤其等位基因的遺傳而得到。人類基因組中大約有100000個RFLP,但是理所應當的每個RFLP只能有兩種等位形式(有或沒有這個位點),這就限制了RFLP在人類基因作圖上的套用價值,因為一個家庭的所有成員很可能都是某一個RFLP的純合子。(簡單來說,它不能區分純合子和雜合子)。
隨機擴增片段長度多態性標記
(Random Amplified Polymorphic DNA ,RAPD)RAPD技術是由Williams等首先創立的一種DNA分子標記技術,利用單一的10個鹼基
寡核苷酸 作為引物,對基因組DNA進行PCR擴增。經瓊脂糖凝膠電泳來檢測DNA序列多態性。
擴增片段長度多態性
(Amplified Fragment Length Polymorphism ,AFLP)AFLP是Zeabeau 等(1993)發明的一項技術,它既有RFLP的可靠性,又有RAPD的方便性。其基本原理是通過PCR 擴增基因組DNA片段,擴增產物的變性
聚丙烯醯胺 電泳顯示擴增片段長度多態性,其中引物=接頭+酶切位點+2~3個核苷酸。
AFLP技術分析流程:⑴DNA 模板製備;⑵提取樣本DNA 經濃度和質量檢測後,一般採用雙酶(EcoRI和MSEI或PstI或TaqI)酶切, 在基因組DNA上產生低頻和高頻切口;⑶選擇性擴增酶切片段。酶切後,限制性片段在T4連線酶作用下與特定接頭連線,形成帶有接頭的特異性片段;⑷PCR前擴增,一般用帶一個選擇性引物進行預擴增,反應條件與常規PCR反應基本一致;⑸利用
放射性同位素 標記或螢光標記PCR 引物;⑹在Taq
聚合酶 作用下完成94 ℃變性30s ,65 ℃淬火30s,72 ℃延伸60s,PCR擴增36個循環;⑺PCR產物在含
尿素 聚丙烯醯胺上電泳;⑻將電泳後的凝膠轉移到吸附濾紙上,經乾膠儀進行乾膠處理;⑼在X光片上感光,數日後沖洗膠片並進行結果分析。
AFLP反應起始一般高溫復性(一般65 ℃),因此只有那些與3’端嚴格配對的片段才能得到擴增,選擇性很強。實驗結果穩定,重複性好,呈典型的孟德爾式遺傳,每個AFLP可以獲得50~100條譜帶信息,多態性強。
微衛星
(Microsatellite)微衛星是指以幾個(1~6 bp) 核苷酸為單位,多次
串聯重複序列 ,也稱之為
簡單重複序列 (single sequence repeats ,SSR) 、
短串聯重複序列 (short tandem repeats ,STR)或簡單序列長度多態性(single sequence length polymorphism ,SSL P)。廣泛分布於
真核生物 基因組中,大約每隔10~50bp就有一個微衛星,由於重複次數和重複程度的不完全而造成每一個位點的多態性。
單核苷酸多態性
(Single Nucleotide Polymorphism,SNP)基因組中存在單個的點
突變 ,且數量極大,有些也對產生RFLP,但許多並個能。這是囪為它們所處的序列不能被限制件內切核酸酶所識別。在人類基因組中,據認為有200000個以上的
SNP (single nucleotidc polymorphism)位乾基因內.而且有更多的SNP位於非基因的DNA中。
每個SNP只有兩個等位基因,所以這些標記在人類繪製
遺傳圖譜 方面又與RFLP同樣的缺點:對於一個SNP,很可能—個家族的所有成員都是純合子。SNP的優點是它數目龐大,而且對SNP分型所用的方法不需
凝膠電泳 。這非常重要,因為已證明凝膠電泳很難實現自動化,所以使用它的檢測方法相對緩慢而且費力。由於SNP以寡核苷酸雜交分析(oligonucleotide hybidization analysis)為基礎,故其檢測更快速。寡核苷酸是在試管中合成的通常小於50個核苷酸的短單鏈DNA分子。在適當的條件下,一個寡核苷酸與另一個DNA分子僅在可形成完全的鹼基配對結構時才能雜交。如果有一個錯配,寡核昔酸上就有一個位置不能形成鹼基對。則不能雜交(圖1)。因此寡核苷酸雜交能區分一個SNP的兩個等位基因。篩選策略包括:
DNA晶片(DNA chip)技術
DNA晶片是一塊面積為2cm平方或更小的矽片,以高密度排列方式攜帶有許多不同的寡核苷酸。待測DNA用螢光標記後加到晶片的表面。用
螢光顯微鏡 觀察雜交情況,顯示螢光信號的位置即表示該處的寡核苷酸與待測DNA發生了
雜交 。因此一個實驗中可以量化許多SNP。
動態等位基因特異的雜交(dynamic allele-specific hybridization,DASH) 在這種技術中,雜交在溶液中進行,比如在96孔微量滴定板的一個孔中進行G螢光標記只能與雙鏈DNA結合,因此只有發生了雜交才能檢測到信號。開始,雜交在允許有錯配的條件下進行。在這個階段,無論待測DNA含有哪一種SNP等位基因,寡核苷酸都能與之雜交。由於錯配的雜交產物不如完全雜交產物穩定,故在較低溫度
解鏈 .因此可以通過升高溫度來區分等位基因。這樣就可以從使雜交依賴性螢光信號消失的溫度來判定待測DNA中存在哪一種等位基因。
備註 並發率和干涉
並發與干涉是影響作圖的因素之一,如果在二條非姊妹染色單體上分別發生兩次
單交換 ,單交換Ⅰ的機率為α,單交換Ⅱ的機率為β,如果這兩次交換同時發生(即為
雙交換 ),其機率理論上為(α)(β),假設實際測得雙交換的機率為γ。
並發係數 (coefficidence of coincidence or coincidence)C為觀察到的雙交換數除以預期雙交換這一比值。
按上列數據
C=γ∕(α)(β)
這個結果遠小於1,也就是說實際發生的雙交換比預期發生的機率要小得多,這是由於一次重組事件的發生,干擾了另一次重組事件的發生,這一現象稱為染色體的干涉(chromasomal interference)或稱為交叉干涉(chiasma interference)。以I來表示。干涉和並發係數的關係以下式表示 I=1-C
當 C=1,I=0時, 表示無干涉存在
C=0,I=1時, 表示存在完全乾涉
1>C>0時 表示存在
正干涉 (positive interference),即是一次交換抑制了鄰近位點另一次交換的發生。這可能是由於染色單體的斷裂和重接物理性地干涉了鄰近位點的交換。
C>1,I<0時,表示存在
負干涉 (negative interference),負干涉僅在
微生物 中發生基因轉變時才出現。
 遺傳作圖
遺傳作圖 遺傳作圖
遺傳作圖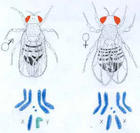 遺傳作圖
遺傳作圖
 遺傳作圖
遺傳作圖 遺傳作圖
遺傳作圖 遺傳作圖
遺傳作圖