背景,長鏈非編碼RNA(lncRNA, lincRNA)資料庫,miR2Disease資料庫,數據蒐集以及資料庫內容,用戶界面介紹,搜尋頁面,提交頁面,討論,失調的microRNA,可能導致microRNA失調的機制,未來的發展方向,
背景
對 RNA進行測序一直以來都被認為是一種發現基因的有效方法,而且這種方法還被認為是對編碼基因以及非編碼基因進行注釋的金標準。與以前的方法相比,大規模 平行RNA測序方法(massively parallel sequencing of RNA)極大增強了RNA測序技術的處理能力,使我們得以能夠對轉錄組進行測序。在本文中即將介紹到的這兩種RNA測序方法就能以前所未有的精度對轉錄組 進行分析。Trapnell小組使用的方法是一種名為Cufflinks的軟體。這種軟體能夠隨時發現小鼠生肌細胞(myoblast cell)內新出現的轉錄子,還能在細胞分化時對轉錄子表達水平進行監測,從而分析基因表達情況和剪接情況。Guttman小組也使用了與 Trapnell小組相類似的軟體方法,不過他們使用的是另一種名為Scripture的軟體。Scripture軟體可以對源自三個小鼠細胞系的轉錄組 進行再注釋(reannotate),從而對數百個最近新發現的lincRNA(large intergenic noncoding RNA)進行完整的基因模式注釋。
雖然RNA測序技術已經出現了將近20年,但直到最近才開始構建克隆文庫。對人類、小鼠以及其它重要模 式生物進行全長基因克隆構建的科研項目需要幾年的時間才能夠完成。但是有了最新的測序技術,我們將不再需要構建克隆文庫,可以直接對cDNA片段進行測 序。我們現在可以只需要花費幾天,僅用以往同類項目科研經費的很少一部分就能夠得到一個比較滿意的完整的細胞轉錄組。但是這種新技術也存在一點問題。不用 構建克隆,我們就無法知道哪一個“結果(mRNA或蛋白)”來自哪一個轉錄子。最近已經有人開始通過對已知的或者預測出來的轉錄子的短RNA序列進行測序 的方式來對基因表達和可變剪接進行分析研究。雖然這些研究可以得到很多信息,但是這種方法只能用於分析已知基因和對已知的可變連線區域進行分析。為了充分 利用RNA序列數據進行生物學研究,我們還應該能夠重建轉錄子並且還要能夠在不藉助參考注釋基因組信息的情況下對這些轉錄子的相對豐度進行精確的測量。
長鏈非編碼RNA(lncRNA, lincRNA)資料庫
國內外長非編碼RNA的研究剛剛興起,種類大致包含以下幾種。
(1)lncRNABase:提供miRNA調控長非編碼RNA(lncRNA)、假基因(pseudogene)和環狀RNA(circRNA)的互作信息和ceRNA調控網路。構建了最全面的包含了14癌症類型(>6000個樣本)Pan-Cancer(泛癌)表達圖譜和互作網路。這些調控互作網路信息是基於高通量的CLIP-Seq實驗數據。
(2)LNCipedia: 對人類的長鏈非編碼RNA的序列和結構全面的注釋。
(3)ChIPBase:提供長鏈非編碼RNA的表達圖譜和轉錄調控的全面鑑定和注釋。整合了高通量的RNA-seq鑑定的lncRNA及其表達圖譜和ChIP-Seq實驗技術鑑定的轉錄因子結合位點。
(4)oncoNcRNA:研究各類非編碼RNA(lncRNA,miRNA, piRNA, tRNA和snoRNA等)在腫瘤中的表達模式和作用。這平台包含了64種癌症類型(>20000腫瘤樣本和>900種癌症細胞系)的各類非編碼RNA的表達和拷貝數變化數據(SCNA)。
(5)lncRNAdb: 提供有生物學功能的長鏈非編碼RNA的全面注釋。這是長鏈非編碼RNA研究領域的大牛John mattick實驗室構建的網站。
(6)LncRNADisease: 提供了文獻報導的疾病相關的長鏈非編碼RNA的注釋。
(7)NONCODE:提供對長鏈非編碼RNA的全面注釋,包括表達和該團隊開發的ncFANs計算機軟體預測的lncRNA功能。這是非編碼RNA研究的知名資料庫,已經更新到第三版。
(8)NRED: 提供人和小鼠的長鏈非編碼RNA在晶片數據的表達信息。這也是John mattick實驗室構建的網站。
miR2Disease資料庫
miR2Disease資料庫是一個人工注釋的資料庫,主要收錄的是與人類疾病相關的microRNA信息,旨在為全世界的科研工作者提供一個全面的與疾病相關的RNA數據資源平台。
miR2Disease資料庫是免費使用的,可以登入網頁www.miR2Disease.org了解更多信息。
miRBase資料庫是一個提供包括miRNA序列數據、注釋、預測基因靶標等信息的全方位資料庫。作為存儲miRNA信息最主要的公共資料庫之一,miRBase提供了便捷的網上查詢服務,允許用戶使用關鍵字或序列線上搜尋已知的miRNA和靶標信息。但它還存在著一些不足的方面,比如植物預測靶標的描述比較模糊、莖環結構顯示方式不夠直觀、特定miRNA相關功能描述的缺乏和對具反饋調節功能miRNA標註的缺乏等問題。希望在不久的將來,隨著miRNA研究的不斷深人,miRBase也建設成一個功能強大的全方位綜合性資料庫。
數據蒐集以及資料庫內容
miR2Disease資料庫中的記錄主要提供的是microRNA失調與人類疾病的關係。他們使用諸如“microRNA disease”、“miRNA disease”、“microRNA cancer”、“miRNA cancer”等關鍵字搜尋了PubMed資料庫。最新版的miR2Disease資料庫是在工作人員瀏覽了600多篇文獻的基礎上總結出來的,共收錄有299個人類microRNA信息、94種人類疾病信息以及1939條microRNA與疾病之間的關聯信息。
在miR2Disease資料庫中他們使用的是由專業的醫學辭彙(即UMLS系統,參見http://diseaseontology.sourceforge.net/)來命名的疾病名稱,這樣便於用戶搜尋和分析資料。資料庫中的每一條記錄都包含詳細的microRNA與疾病之間的關聯信息、microRNA ID號、相關疾病名稱、患病時microRNA表達模式(上調還是下調)介紹、microRNA表達模式相關試驗技術介紹(晶片、northern blot、qRT-PCR等)、microRNA靶基因介紹(這些基因都是參閱相關文獻或者直接從TarBase資料庫中查閱而來)。
miR2Disease資料庫使用的界面非常人性化,用戶可以很方便地使用microRNA ID號、相關疾病名稱或者靶基因名稱進行查詢操作,而且還提供了許多外部microRNA資料庫的連結,例如microRNA序列及注釋信息資料庫miRBase、經過試驗驗證的microRNA靶基因資料庫TarBase,以及microRNA功能計算機預測資料庫TargetScan、miRanda和PicTar。
此外,miR2Disease資料庫還提供與NCBI PubMed中參考文獻之間的超連結及該文獻在PubMed中的ID號和完整的引文介紹。在最新版本的miR2Disease資料庫中,有1/7的記錄是有關microRNA失調導致人類疾病相關信息的。
用戶界面介紹
搜尋頁面
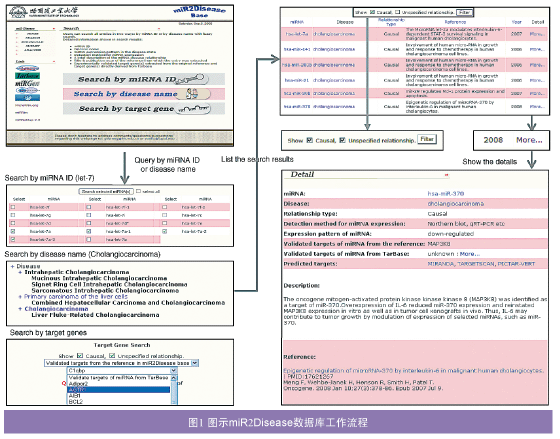
miR2Disease資料庫可以通過搜尋引擎在庫中查詢microRNA——疾病相關信息,用戶可以通過microRNA ID號、相關疾病名稱或者靶基因名稱進行查詢。
miR2Disease資料庫還提供一種模糊查找功能,用戶即使不知道疾病的確切名稱,也可以藉助該功能通過醫學疾病參考辭彙進行查詢。
用戶輸入一個查詢關鍵字之後,系統就會在資料庫中搜尋出所有包含該關鍵字的記錄,這些記錄會以“疾病樹(disease tree)”的形式展現出來,其中既包含了疾病本身,也包含其“祖先節點(ancestor node)”和所有的亞類。在“疾病樹(disease tree)”中包含查詢關鍵字的部分會以高亮粗體的方式顯示出來,每一個包含有microRNA——疾病相關信息的部分也會給出超連結,用戶通過這些超連結就可以很輕鬆地獲得與該疾病相關的所有microRNA的信息。在搜尋結果頁面,點擊每一條記錄末尾處的‘more . . .’按鈕可以了解到更多相關的信息(如圖)。
與使用疾病名稱進行搜尋差不多,使用microRNA名稱進行搜尋也有可能出現混淆與混亂,因為大部分的原始文獻都不會給出某一個microRNA家族中每一個具體microRNA的詳細信息及名稱。例如在很多文獻中提到的“let-7”是與腺瘤相關的,但還有一些文獻中則報導說在乳腺癌患者體內let-7a-3的表達水平會降低。因此,通過模糊查詢功能,用戶哪怕在不清楚microRNA準確名稱的情況下也能在miR2Diesease資料庫中查詢到相關信息。
有時使用一個microRNA名稱可以查到好幾個ID號,用戶可以根據自己的興趣做進一步選擇。圖1也給出了使用miRNA ID號進行查詢的簡要流程圖。
在miR2Disease資料庫中,
靶基因可以被分為三類,即原始
參考文獻中報導的靶基因;TarBase資料庫(該資料庫收錄的都是經過試驗驗證的數據)報導的靶基因;以及使用計算機軟體(例如Miranda、TargetScan、PicTar)預測出的靶基因。用戶可以使用第一和第二種靶基因進行搜尋,目前還不支持使用第三種靶基因進行搜尋,不過今後的版本肯定能支持該功能。
microRNA與疾病的關係在搜尋結果頁面和詳細介紹頁面都有顯示(圖1)。而且,miR2Diesease資料庫還提供過濾功能,用戶可以選擇只顯示microRNA與疾病關係的相關信息。
提交頁面
miR2Disease資料庫還設有用戶提交頁面,供用戶上傳目前miR2Disease資料庫中還沒有收錄的有關microRNA與人類疾病關係的相關數據資料。一旦miR2Disease
資料庫評審委員會審核通過用戶提交的信息,資料庫就會將該信息收錄進來,在接下來的新版本中公布。miR2Disease資料庫會每月更新一次。
討論
越來越多的證據表明,某種microRNA時空特異性的表達對於細胞正常的發育分化過程起到了至關重要的作用,而microRNA的異常表達則與許多人類疾病有關。為了給研究microRNA與人類疾病關係的科學家們提供一個集中的相關數據資源,研究人員有了建立miR2Disease資料庫的想法。他們主要關注microRNA失調與人類疾病的關係。
miR2Disease資料庫不僅僅是一個供用戶查詢信息的
資料庫,同時它也是一個可以上傳資料,供大家交流信息、分享成果的平台。
失調的microRNA
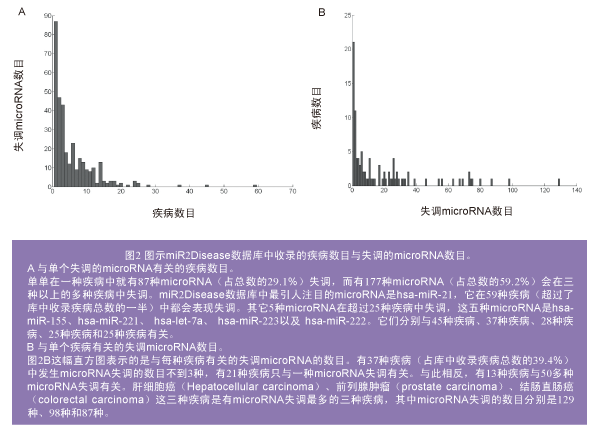
在miR2Disease資料庫收錄的條目中大約有1/7的microRNA失調導致疾病發生,它們會引起癌症、代謝性疾病、心血管疾病等等。例如Ma等人報導的miR-10b高表達抑制了HOXD10基因的翻譯,引起RHOC這種促轉移基因(pro-metastatic gene)高表達,從而導致乳腺癌擴散、轉移。再如Huang等人發現miR-373和 miR-520c的明顯上調抑制了CD44表達,因而刺激了乳腺癌細胞的轉移。Meng等人則發現癌基因——促分裂原活化
蛋白激酶激酶(mitogen-activated protein kinase kinase kinase 8, MAP3K8)是miR-370的
靶基因,miR-370下調會導致MAP3K8基因表達水平升高,引起膽管癌(cholangiocarcinoma)。還有報導miR-375通過調控肌侵蛋白(myotrophin)的表達能控制胰島素分泌,miR-375上調能抑制胰島素釋放。還有研究表明miR-1和miR-133下調與HCN2/HCN4再表達(reexpression)和肥大心肌細胞電重構(remodeling)等有關。
miR2Disease資料庫中的信息還可以用來研究不同疾病之間的關係,他們為此專門設立了一個雙向網路(bipartite network),以描述85種microRNA和32種癌症相關疾病之間的因果關係(圖3)。
可能導致microRNA失調的機制
首先,microRNA都是位於與疾病有關的
基因座內,例如雜合子缺失的微小區域(minimal regions of loss of heterozygosity)、微小擴增區域(minimal amplicon)或斷裂位點(breakpoint cluster region)等脆性位點區域。比如miR-15和miR-16都位於人染色體13q14區域,該區域在超過半數的B細胞性慢性淋巴細胞性白血病(B-cell chronic lymphocytic leukemia,B-CLL)患者中都出現缺失。因此,大多數的B-CLL患者(68%)中這兩個基因也都有缺失或表達下調。相反,miR-17-92多順反子位於B細胞淋巴瘤基因擴增區域,因此會過表達。
其次,microRNA失調是由異常的表觀遺傳學改變(epigenetic patterns)所致。例如DNA異常甲基化、組蛋白異常修飾等等。比如,在正常情況下,人類正常組織中let-7a-3基因的啟動子區域是高度甲基化的,但是在肺癌組織中該區域則是低甲基化的。這種啟動子低甲基化狀態會引起let-7a-3這種癌基因激活,引發肺癌。此外,異常的高甲基化會導致乳腺癌患者體內miR-9-1失活。
最後,microRNA失調可能是由參與microRNA生成的酶功能異常所致。比如,Otsuka等人發現的miR-24和miR-93可以針對病毒L蛋白(large protein)和P蛋白(phospho protein)基因。在Dicer1缺陷的細胞中,缺乏miR-24和miR-93會增強VSV病毒的複製效率。在miR2Disease資料庫中,上述這些信息都位於“分析類”中,可以從網站主頁上連結進入。
除了上述內容之外,miR2Disease資料庫中還收錄有關哪些導致疾病的
基因變異位點能通過干擾microRNA的靶向效應來影響細胞功能的條目。例如,let-7:Hmga2、miR-148a:HLA-G和miR-433:FGF20之間的相互靶向作用消失分別與肌瘤(myoma)、哮喘(asthma)以及帕金森氏症(Parkinson’s disease)相關。
Chen等人還發現CCND1 mRNA通常都會受到miR-16-1的調控,而在外套細胞淋巴瘤(mantle cell lymphoma)細胞中,CCND1基因的截短突變體則不受miR-16-1的調控,因為該突變體基因的mRNA上沒有miR-16-1的結合位點。總之,microRNA不能對靶基因進行調控(microRNA與mRNA之間失去了相互作用的能力或不能互相結合等)是microRNA致病的一條新機制。
綜上所述,miR2Disease資料庫為人們提供了一個有關microRNA失調與人類疾病關係方面的綜合網上數據資源平台。有理由相信miR2Disease資料庫會成為生命科學和生物學界研究工作者的好幫手,能幫助他們進一步了解microRNA失調與人類疾病之間的關係。
未來的發展方向
由於miR2Disease資料庫的主要工作是“數據收集與整理、儲存”,因此,研究人員目前主要是使用一些諸如“microRNA disease”、 “miRNA disease”、 “microRNA cancer”、“miRNA cancer”等關鍵字在PubMed資料庫中進行有關microRNA與人類疾病關係方面的資料蒐集整理工作。雖然到目前為止研究人員已經從600多篇文獻中蒐集到了約2000條microRNA與人類疾病的關係並收錄入miR2Disease資料庫中,但是由於存在上述建庫方法學上的局限性,因此,miR2Disease資料庫目前還缺乏全面性與系統性。所以他們計畫採用兩種新的策略來改進採集數據的方法。第一條策略是將使用文本挖掘工具(text-mining tool)來幫助對PubMed資料庫的摘要進行預檢索(prescreen),以此來擴大初步搜尋的範圍。第二條策略是將重點關注美國國家醫學圖書館(National Library of Medicine)建立的MeSH辭彙表更新情況。
毫無疑問,上述這兩條策略將豐富miR2Disease的數據量,擴大資料庫的覆蓋範圍,相信在即將發布的新版miR2Disease資料庫中,用戶就會看到這些改變了。
值得注意的是不是所有的microRNA與疾病之間都有非常明確的關係,miR2Disease資料庫中有一些記錄比其它的記錄有更明確的microRNA——疾病關係。