醫學圖像分析(Medical Image Analysis)是醫學術語,指綜合醫學影像、數學建模、數字圖像處理與分析、人工智慧和數值算法等學科的交叉領域。
基本介紹
- 中文名:醫學圖像分析
- 外文名:Medical Image Analysis
醫學圖像分析簡介
理論
分割
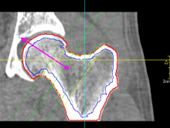
圖像配準和信息融合
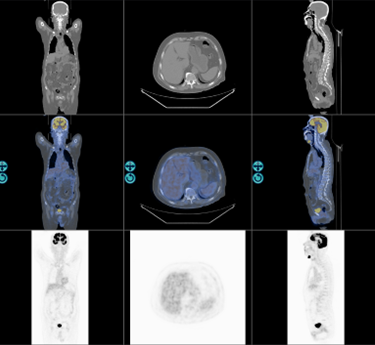
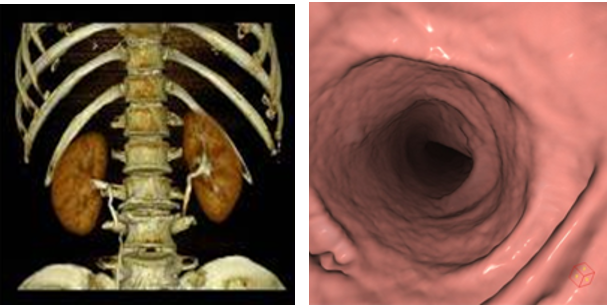
可視化
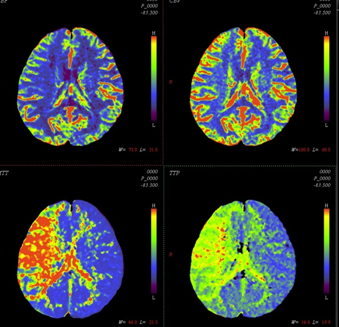
時序圖像的功能分析
功能拓展及套用
擴展閱讀
- 陳武凡, 秦安, 江少峰, 馮前進, 郝立巍. 醫學圖像分析的現狀與展望, 《中國生物醫學工程學報》,2008年第27卷第2期 175-181頁。
- 湯敏, 陳峰, 陶玲. 醫學圖像可視化及加速技術的研究進展和趨勢, 《中國組織工程研究與臨床康復》,2011年第15卷第4期 741-744頁。
- J Gee, M Reivich, R Bajcsy (1993). "Elastically Deforming a Three-Dimensional Atlas to Match Anatomical Brain Images". Journal of Computer Assisted Tomography 17 (1): 225–236.
- MR Sabuncu, BT Yeo, K Van Leemput, B Fischl, P Golland (June 2010). "A Generative Model for Image Segmentation Based on Label Fusion". IEEE Transactions on Medical Imaging 29 (10): 1714–1729.
- Cootes TF, Taylor CJ, Cooper DH, and Graham J (1995). "Active shape models-their training and application". Computer vision and image understanding (San Diego: Academic Press, c1995-) 61 (1): 38–59.
- Cootes, T.F. and Edwards, G.J. and Taylor, C.J. (2001). "Active appearance models". Pattern Analysis and Machine Intelligence, IEEE Transactions on (IEEE) 23 (6): 681–685.
- R. Goldenberg, R. Kimmel, E. Rivlin, and M. Rudzsky (2001). "Fast geodesic active contours". IEEE Transactions on Image Processing 10 (10): 1467–1475.
- Karasev, P., Kolesov I., Chudy, K. Vela, P., and Tannenbaum, A. (2011). "Interactive MRI segmentation with controlled active vision". Proceedings of the Conference on Decision and Control.
- Lisa Gottesfeld Brown (1992). "A survey of image registration techniques". ACM Computing Surveys (CSUR) 24 (4): 325–376.
- J. Maintz and M. Viergever (1998). "A survey of medical image registration". Medical Image Analysis 2 (1): 1–36.
- J. Hajnal, D. Hawkes, and D. Hill (2001). Medical Image Registration. Baton Rouge, Florida: CRC Press.
- Barbara Zitová, Jan Flusser (2003). "Image registration methods: a survey". Image Vision Comput. 21 (11): 977–1000.
- J. P. W. Pluim, J. B. A. Maintz, and M. A. Viergever (2003). "Mutual information based registration of medical images: A survey". IEEE Trans. Med. Imaging 22 (8): 986–1004.
- C. J. Twining and T. Cootes and S. Marsland and V. Petrovic and R. Schestowitz and C. Taylor. "A unified information-theoretic approach to groupwise non-rigid registration and model building". Proceedings of Information Processing in Medical Imaging 2005.
- M. Sabuncu and S. K. Balci and M. E. Shenton and P. Golland (2009). "Image-driven Population Analysis through Mixture Modeling". IEEE Transactions in Medical Imaging 28 (9): 1473–1487.
- K.J. Friston, W.D. Penny, C. Phillips, S.J. Kiebel, G. Hinton, and J. Ashburner (2002). "Classical and Bayesian Inference in Neuroimaging: Theory". NeuroImage 16 (2): 465–483.
- Yong Fan, Nematollah Batmanghelich, Chris M. Clark, Christos Davatzikos, the Alzheimer’s Disease Neuroimaging Initiative, Spatial patterns of brain atrophy in MCI patients, identified via high-dimensional pattern classification, predict subsequent cognitive decline. (2008). "Spatial patterns of brain atrophy in MCI patients, identified via high-dimensional pattern classification, predict subsequent cognitive decline". NeuroImage 39 (4): 1731–1743.
- Y. Wang and Y. Fan and P. Bhatt P and C. Davatzikos (2010). "High-dimensional pattern regression using machine learning: from medical images to continuous clinical variables". Neuroimage 50 (4): 1519–35.
- Benoît Magnin and Lilia Mesrob and Serge Kinkingnéhun and Mélanie Pélégrini-Issac and Olivier Colliot and Marie Sarazin and Bruno Dubois and Stéphane Lehéricy and Habib Benali (2009). "Support vector machine-based classification of Alzheimer's disease from whole-brain anatomical MRI". Neuroradiology 51 (2): 73–83.
- N.K. Batmanghelich and B. Taskar and C. Davatzikos (2012). "Generative-discriminative basis learning for medical imaging". IEEE Trans Med Imaging 31 (1): 51–69.
- Glenn Fung and Jonathan Stoeckel (2007). "SVM feature selection for classification of SPECT images of Alzheimer's disease using spatial information". Knowledge and Information Systems 11 (2): 243–258.
