mRNA差異顯示技術(mRNA differential display PCR, mRNA DD-PCR),是由Peng Liang等人在1992年建立的篩選基因差異表達的有效方法。它是將mRNA逆轉錄技術和PCR技術相結合的一種RNA指紋圖譜技術。每一種組織細胞(包括同一組織細胞經過不同的處理)都有其特異表達的不同於其他組織細胞的基因譜,即特異的RNA指紋圖譜。
基本介紹
- 中文名:mRNA差異顯示技術
- 外文名:mRNA differential display PCR
- 簡稱:mRNA DD-PCR
- 建立時間:1992年
原理
差異基因表達是細胞分化的基礎。正是這些基因在細胞中的特異表達與否,決定了生命歷程中細胞的發育和分化、細胞周期的調節、細胞衰老和死亡等。mRNA DD-PCR技術正是對組織特異性表達基因進行分離的一種快速而行之有效的方法。不同的組織/細胞或遺傳背景相同的同一組織/細胞經過不同的處理後,提取各自的總RNA,進行mRNA的逆轉錄合成 cDNA。進行逆轉錄時3'端引物採用oligo(dT)l2MN,其中M為A、C、G中的任何一種,N為A、C、G或T中的任何一種,所以共有12種oligo(dT)l2MN引物。其申M稱為錨定引物,起增大引物Tm值的作用。N稱為分類鹼基,對逆轉錄進行歸類。
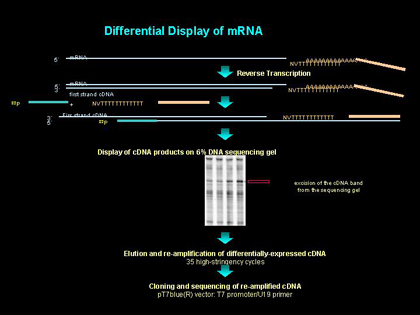
實驗流程
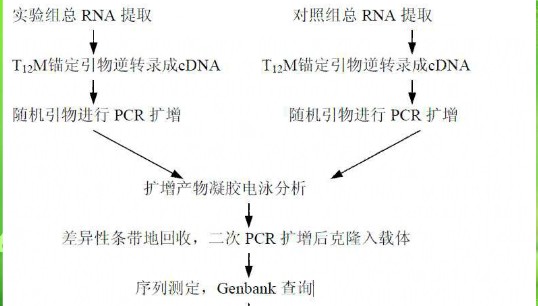
特點
通常情況下,在某一細胞中或某一個關鍵的發育時間點上有15000種以上的基因在表達。如此眾多種類的mRNA逆轉錄產物經PCR選擇性擴增後其類型依然眾多,很難用電泳系統加以快速準確地分離。因而需要對逆轉錄產物的cDNA進行歸類處理,減輕不同PCR產物電泳分離的難度,提高分離的準確率。該方法就具有快速、靈敏和重複性好等優點。
套用
mRNA DD-PCR技術是篩選和克隆差異表達基因的一種快速而行之有效的方法。
