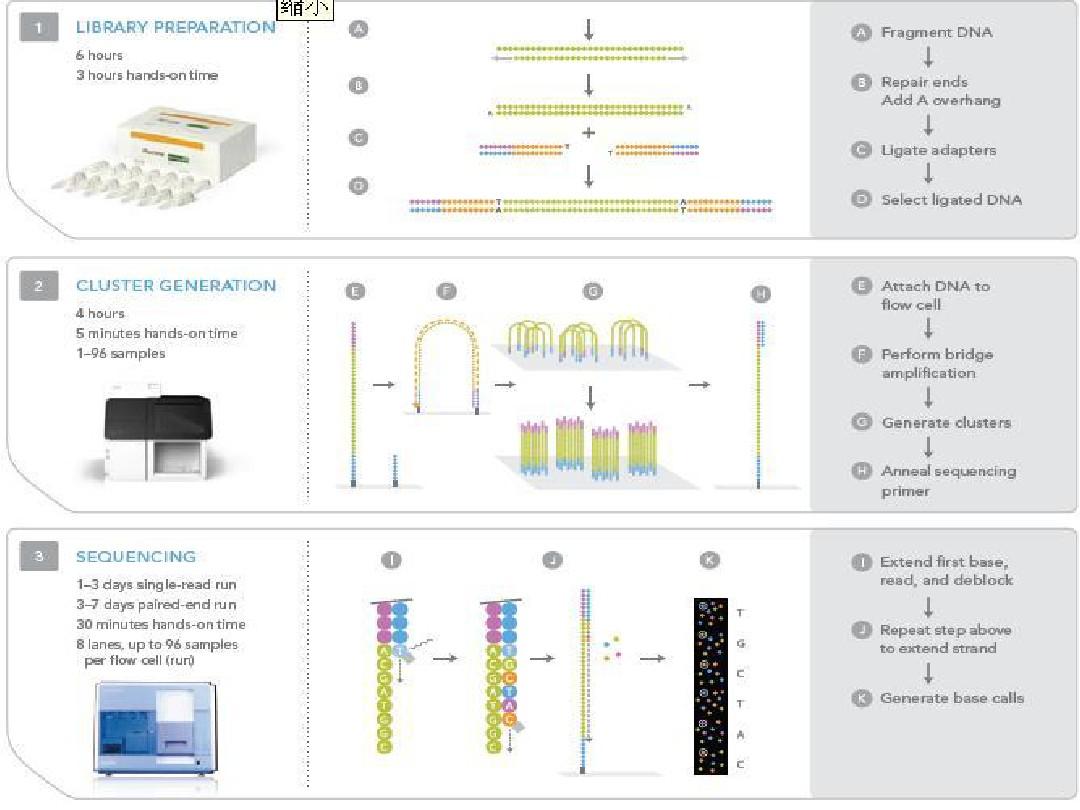
第二代DNA測序技術又稱下一代測序技術,是對第一代測序技術的劃時代變革的核心。現有的技術平台主要包括Roche/454 GS FLX、Illumina/Sol-exa GenomeAnalyzer、Helicos BioSciences公司的HeliScope™ Single Molecule Sequencer、美國Dana-her Motion公司推出的Polonator;以及連線法測序 (sequencing by ligation),即通過引物來定位核酸信息,技術平台有Applied Biosystems/SOLiD™ system。以上技術平台所運用的測序原理均為循環微陣列法。
基本介紹
- 中文名:第二代DNA測序技術
- 外文名:Next-generation sequencing
- 技術平台:Roche/454 GS FLX、Illumina/Sol-exa GenomeAnalyzer等
背景,DNA測序技術,第一代DNA測序,基本平台,454焦磷酸法平台,Solexa基因組分析儀,SOLiD高通量測序儀,HeliScope測序儀,
背景
DNA測序技術
長期以來,DNA測序技術一直是分子生物學相關研究中最常用的技術手段之一,從一定程度上推動了該領域的快速發展。人類基因組計畫、轉錄組分析、微生物基因組重測序、單核苷酸的多態性 (single nucleotide polymorphisms,SNP) 分析等方面也促進的其他生物學領域的研究和發展。每一代測序技術的更替都標誌著生物學中基因晶片、數據分析、表面化學、生物工程等技術領域有了新的突破,從而套用在了測序領域,大大降低了測序成本,提高了測序效率,使測序向著高通量、低成本、高安全性和商業化的方向發展。
第一代DNA測序
儘管第一代 DNA 測序技術以其可達 1000 bp 的測序讀長、99.999% 的高準確性幫助人們完成了大量的測序工作,但其測試速度慢、成本高、通量低等方面的不足,也致使其不能得到大眾化的套用。隨著科學技術的進步以及科研人員對測序技術的努力開發,2005 年 Roche 公司發布的 454 測序系統標誌著測序技術跨人高通量並行測序的時代。第二代 DNA 測序技術又稱大量並行測序技術 (massive parallel sequencing,MPS)、高通量測序技術(high—throughput sequencing,HTS),以低成本、99% 以上的準確度,1次可對幾百、幾千個樣本的幾十萬至幾百萬條 DNA 分子同時進行快速測序分析。這一時期的代表技術有 Roche 公司的 454、Illumina公司的 Solexa、ABI公司的SOLID,由於該時期的測序技術十分前沿,因而市場主要被這三家公司所壟斷。
基本平台
454焦磷酸法平台
454焦磷酸法平台是最早上市的循環微陣列法平台,使用的是邊合成邊測序 (sequencing by synthesis, SBS)技術,避免了Sanger法存在的宿主菌克隆問題。
①首先將待測的目的DNA分子打斷成 300-800 bp 的片段,然後在 DNA 片段的 5′ 端加上一個磷酸基團,3′ 變成平端,在兩端分別加上 44 bp 的 A、B 兩個銜接子,組成目的 DNA 的樣品文庫。加上這樣兩個銜接子 A、B 的目的是為了其在生物素和鏈霉親和素的作用下,同含有過量鏈霉親和素的磁珠特異性結合。
②目的 DNA 片段固定到一個磁珠上之後,將磁珠包被在單個油水混合小滴(乳滴),在這個乳滴里進行獨立的擴增,而沒有其他的競爭性或者污染性序列的影響,從而實現了所有目的 DNA 片段進行平行擴增乳滴 PCR (emul-sion PCR, emPCR),經過富集之後,每個磁珠上都有約 107 個克隆的 DNA 片段。
③隨後將這些DNA片段放入 PTP(Pico TiterPlate) 反應板中進行後繼測序。PTP平板含有 160 多萬個由光纖組成的孔,孔中載有化學發光反應所需的各種酶和底物。
④測序開始時,放置在四個單獨的試劑瓶里的四種鹼基,依照 T、A、C、G 的順序依次循環進入 PTP 板,每次只進入一個鹼基。如果發生鹼基配對,就會釋放一個焦磷酸鹽 (inorganic pyrophosphate, PPi) 分子。PPi 在ATP硫酸化酶的催化下與腺苷醯硫酸反應生成 ATP,ATP 與蟲螢光素反應發光,光信號的最大波長約為 560 nm。
⑤此反應釋放出的光信號實時被儀器配置的高靈敏度 CCD 捕獲到。有一個鹼基和測序模板進行配對,就會捕獲到一分子的光信號,由此一一對應,就可以準確、快速地確定待測模板的鹼基序列,讀長可達到近 500 bp。
Solexa基因組分析儀
Solexa 分析儀通常也被稱為 Illumina 測序儀,所使用的方法是克隆單分子陣列技術。
①將目的DNA分子打斷成 100-200 bp 的片段,隨機連線到固相基質上,經過 Bst 聚合酶延伸和甲酸胺變性的橋PCR循環,生成大量的 DNA簇 (DNA cluster),每個DNA 簇中約有1000個相同序列的 DNA 片段。
②之後的反應與Sanger法類似,加入用4種不同螢光標記並結合了可逆終止劑的 dNTP。固相基質上每個孔有八道獨立檢測的位點,所以一次可以並行八個獨立文庫,可容納數百萬的模版克隆,可把多個樣品混合在一起檢測,每個固相基質上一次可讀取10億個鹼基。
③DNA 簇與單鏈擴增產物的通用序列雜交,由於終止劑的作用,DNA 聚合酶每次循環只延伸一個 dNTP。每次延伸所產生的光信號被標準的微陣列光學檢測系統分析測序,下一次循環中把終止劑和螢光標記基團裂解掉,然後繼續延伸 dNTP,實現了邊合成邊測序技術。
④其主要的缺點是由於光信號衰減和移相的原因使得序列讀長較短,可以進行每個 DNA 測序片段的閱讀長度僅為 75 bp 的末端雙向測序反應。
SOLiD高通量測序儀
SOLiD(sequencing by oligonucleotideligationand detection, SOLiD)測序技術最初是由哈佛大學 Church 研究小組成員Shendure等所發明的。Church 研究組發明的方法的一部分授權給了 Agen-court 公司,並在 2006年7月被美國套用生物系統公司 (Applied Biosystems lnc, ABI) 收購,組建 SOLiD 測序平台。基於該原理的最新測序平台是於2010年末推出的5500xl,已是第五代產品。
①首先製備 DNA 文庫。SOLiD 技術支持兩種測序文庫,分別是片段文庫(fragment library)和配對末端文庫(mate-paired library)。將待測的DNA分子打斷,並在兩端加上接頭,則可組成片段文庫。而配對末端文庫則是先把DNA分子打斷,在中間加入 EcoP15 酶切位點和internal接頭後進行環化,然後用 EcoP15 酶切,使得接頭的兩端各有27 bp的鹼基,最後在兩端加上接頭,構成文庫,適用於全基因組測序、SNP分析、結構重排及拷貝數的分析等相關領域。
②第二個階段與454焦磷酸測序法相同,加入磁珠等反應元件進行 emPCR 平行擴增,不同的是該方法的磁珠只有1 µm。在連線測序中,底物是 8 個鹼基的八聚體單鏈螢光探針,在5′末端分別標記了CY5、Teaxs Red、CY3、6-FAM這四種顏色的螢光染料。3′ 端的第 1、2 位鹼基類別排序分別對應著一個固定的螢光染料,第 3、4、5 位鹼基“n”是隨機鹼基,第 6、7、8 位鹼基“z”是可以和任何鹼基配對的特殊鹼基。
③一次測序中包括了五輪連線反應。每輪連線反應首先是由3個鹼基“n”介導,將八聚體連線在引物上,測序儀記錄螢光染料信號,然後斷裂掉鹼基“z”,準備連線下一個八聚體。一次循環後,將引物重置,進行第二輪連線反應,反應位置比前一輪錯開一位,這樣引物上的每個鹼基都會有兩次與第1、2位的相連線,顯著減小了測序誤差 。
HeliScope測序儀
HeliScope 測序儀是由 Quake 團隊設計開發的,從原理層面上來看,屬於第二代測序原理,是循環晶片測序儀的一種。但是其在第二代的基礎上引入了單分子測序的概念,被稱為2.5代。
①該方法克服了第二代測序技術中需要用 PCR 擴增來增強螢光信號這一技術難題,採用了更加靈敏的圖像感測器 (charge-coupled device, CCD) 可以出檢測單分子 dNTP 攜帶的螢光基團發出的信號,單次掃描時間只有 15 ms 。
②HeliScope利用精密的全內反射顯微鏡 (total internal reflection microscopy) 技術,在測序結束後去掉延伸的合成鏈,從相反的方向再進行另一次測序,生成同一模板的第二個序列信息,雙向測序顯著提高了原始數據準確度 。
③HeliScope 測序儀和 454 測序儀一樣,測序是不同步完成的,而測序速度由每條模版的序列來確定。由於大量的未標記鹼基、不發光鹼基和污染鹼基的滲入,檢測確實突變的出錯率較高,但檢測鹼基替換突變的誤差率較低。